Interne Dynamik im aktiven Zentrum eines starr geglaubten Enzyms
- Neues aus der Fakultät 2020
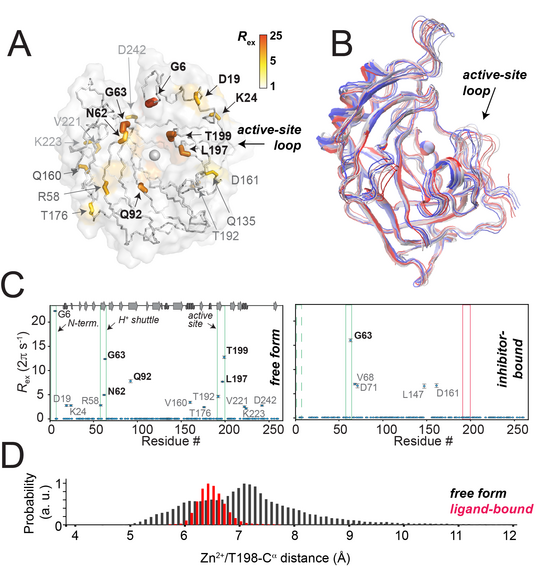
Auf Grundlage der Standardpipeline strukturbasierter Medikamentenforschung sind Wissenschaftler während jahrzehntelangen Gebrauches des Proteins als Modellsystem für medizinische Chemie und Bindungskinetik davon ausgegangen, dass Enzymdynamik für die Katalyse der Familie der Carboanhydrasen und ihre Interaktion mit Inhibitoren keine Rolle spiele. In ihrer Publikation in der Angewandten Chemie zeigen Dr. Himanshu Singh, Dr. Suresh Vasa, Prof. Dr. Rasmus Linser und Kollegen von der Ruhr-Uni Bochum, dass eine konformationelle Dynamik zwischen offener und geschlossener Form existiert, die auf derselben Zeitskala liegt wie die Biokatalyse des Enzyms selbst. Der Fund, der aus detaillierten Relaxationsdispersionsexperimenten und Vergleich dieser Daten in kristalliner und nativer, gelöster Form des Proteins herrührt, wird in der gerade erschienenen Arbeit mechanistisch durch den Einsatz von Molecular Dynamics Studien der Gruppe Schäfer an der RUB komplettiert, die im Rahmen des Exzellenzclusters RESOLV mit der TU Dortmund interagiert
Als eines der absoluten Standardsysteme im Bereich der medizinischen Chemie und vielfach verwendetes Lehrbuchbeispiel mit über 700 Kristallstruktureinträgen steht das Protein stellvertretend für eine Vielzahl von Proteinen, deren Dynamik durch das normalerweise auf Kristallisation und kryogene Temperaturen ausgelegte Arsenal des structure-guided Drug Designs konsequent übersehen wird, was die Interpretation für die kinetischen Parameter der Inhibition schwierig macht und Verbesserungspotenzial für das Feintuning während der Schrittes der Lead Optimization aufzeigt.