Aktivierung eines Rezeptors durch allosterische Regulation eines dynamischen konformationellen Ensemble
- Neues aus der Fakultät 2025

Aufgrund der starken Zunahme von bakteriellen Antibiotikaresistenzen nimmt das Interesse an bakterielle Signalkaskaden stetig zu. Zyklisches Diadenosinmonophosphat (c-di-AMP) ist ein wichtiger bakterieller sekundärer Botenstoff, der unter anderem in Wachstum, Bildung von Biofilmen, Virulenz und Genexpression involviert ist. Die Aktivierungsmechanismen von c-di-AMP-Rezeptoren, wie dem 44 kDa schweren, homotrimeren PII-ähnlichen Protein A (PstA), infolge von Botenstoffbindung blieben jedoch aufgrund der zentralen Rolle von hoch-flexiblen Proteinregionen bisher schwer fassbar. Da für die verwandte Klasse an PII-Signaltransduktionsproteinen bekannt ist, dass die trimeren PII-Proteine durch drei hervorstehende, flexible Loops an trimere Zielproteine wie Amphotericin B (AmtB) und N-Acetylglucosaminkinase (NAGK) binden können, wird solch eine Signalweiterleitung auch für PstA vermutet. Abgesehen von den an das Zielprotein gebundenen Komplexen bleiben die ausgedehnten Loops aller PII- und PII-ähnlicher Proteine in Röntgenkristallstrukturen größtenteils unsichtbar und die wenigen beobachtbaren Strukturmerkmale müssen aufgrund von intermolekularen Kristallkontakten mit Vorsicht behandelt werden.
Das homotrimere PstA besitzt einen Kern mit einer doppelten βαβ-ferrodoxinähnlichen Faltung, wobei die verschiedenen Sekundärstrukturelemente durch einen kurzen T-Loop an der Ligand-Bindungstasche und einem ausgedehnten, flexiblen B-Loop verbunden sind (vgl. Abb. 1). Beide Loops sind in der Ligand-ungebundenen Form in der Röntgenkristallstruktur unsichtbar. Nach Ligandbindung zeigt der kurze T‑Loop eine wohldefinierte Elektronendichte, während der Kern des Proteins eine nahezu identische Struktur aufweist und der B‑Loop weiterhin unsichtbar bleibt, was die Frage aufwirft, wie Ligandbindung eine erhöhte Bindungskompetenz zu nachgeschalteten Interaktionspartnern verursachen kann.
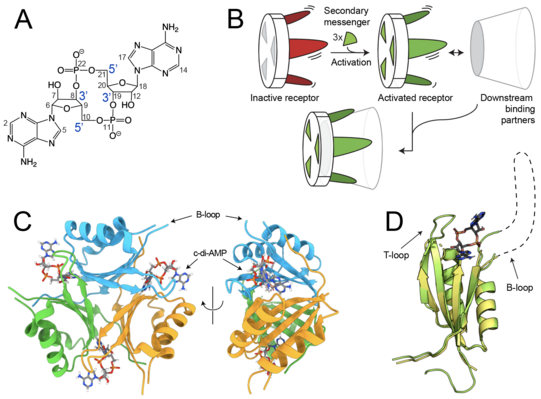
Im Gegensatz zur Röntgenkristallographie und Kryoelektronenmikroskopie ermöglicht NMR-Spektroskopie hingegen die Messung von Dynamiken auf verschiedenen Zeitskalen. Mithilfe von Relaxationsexperimenten, die Dynamiken auf der langsamen µs-ms Zeitskala charakterisieren, konnte in der Ligand-ungebundenen Form für die beiden an den T-Loop gebunden β-Faltblätter im rigiden Kern des Proteins ein konformationeller Austausch auf der µs-Zeitskala nachgewiesen werden. Diese unerwartete Plastizität des Kerns könnte die Aufnahme des c-di-AMP-Liganden, der in röntgenkristallographischen Studien als tief im Komplex vergraben gefunden wurde, deutlich erleichtern, während sie im Falle eines komplett starren Kerns kinetische gehindert wäre. Verschiedene Relaxationsexperimente auf der schnellen ps-ns Zeitskala zeigten eine Zunahme der Unordnung in Richtung des B-Loop-Zentrums, wobei eines der Experimente in der Apoform auf einen zusätzlichen Austauschbeitrag des B-Loops auf einer langsameren Zeitskala hindeutete. Die Relaxationsmessungen sind daher weder mit sich von Botenstoffbindung völlig unbeeinflusst bewegenden B-Loops, noch mit wohldefinierten Loop-Konformationen vereinbar. Stattdessen deuten die NMR-Experimente auf ligandbindungsabhängige Unterschiede der transienten Interaktionen zwischen den dynamischen Loops und dem Rezeptorkern hin, wodurch die räumlichen Eigenschaften des Loops empfindlich verändert würden und eine nachfolgende Signalweiterleitung über die erleichterte Bindung von Zielproteinen ermöglicht würde.
Um die experimentell beobachteten Unterschiede im Loop-Verhalten mit mehr mechanistischen Details zu ergänzen, wurden Moleküldynamiksimulationen (MD Simulationen) durchgeführt. Sechs auf verschiedenen Startstrukturen basierende, 1 µs lange MD-Replikasimulation (enstpricht einer Simulationszeit von 18 µs bezüglich des für den B‑Loop gesampelten Raums) für die apo- und c-di-AMP-gebundene Form zeigten, dass Ligandbindung durch eine Vielzahl neu gebildeter und stabilisierter transienter Kontakte zwischen B‑Loop, Kern, T-Loop und Ligand den konformationellen Raum des B‑Loops in Richtung hin zu einem potentiellen Zielproteins einschränkt, während gleichzeitig ein hoher Grad an lokale Flexibilität des Loops erhalten bleibt (vgl. Abb. 2). Durch eine Quantifizierung der kollektiven Ausrichtung aller drei B‑Loops hin zum potentiellen Bindungspartner konnte gezeigt werden, dass Ligandbindung zu einer mehrfach erhöhten Anzahl an bindungskompetenten Trimerkonformationen führt, was vor allem im Hinblick auf die in der Zelle niedrigen Rezeptorkonzentrationen einen entscheidenden kinetischen Vorteil zur Signalweiterleitung darstellen könnte.

Benedikt Söldner, Himanshu Sigh, Elias Akoury, Gregor Witte, Rasmus Linser,
Angew. Chem. Int. Ed. 2025, e202509394, DOI: 10.1002/anie.202509394
Benedikt Söldner, Himanshu Sigh, Elias Akoury, Gregor Witte, Rasmus Linser,
Angew. Chem. 2025, e202509394, DOI: 10.1002/ange.202509394






