Archiv 2022
Mit hochdimensionaler Festkörper-Kernmagnetresonanzspektroskopie zu neuen Erkenntnissen über große Enzymkomplexe
- Neues aus der Fakultät 2022
Protein-Kernmagnetresonanzspektroskopie (NMR) ist eine weit verbreitete Methode, um die Struktur und Dynamik von Proteinen aufzuklären. NMR kann dabei mit atomarer Auflösung in das Innere von Proteinen schauen. Da sie auch Zugang zu Wasserstoffatomen (genauer, zu Protonen) liefert, ist die NMR eine wertvolle Ergänzung zu anderen strukturbiologischen Techniken wie Kryo-Elektronenmikroskopie (cryo-EM) und Röntgenstrukturanalyse. Die protonendetektierte Festkörper-NMR hat dabei den speziellen Vorteil, Moleküle prinzipiell unabhängig von ihrem Molekulargewicht studieren zu können, ein Wettbewerbsvorteil gegenüber der weiter verbreiteten Lösungs-NMR.

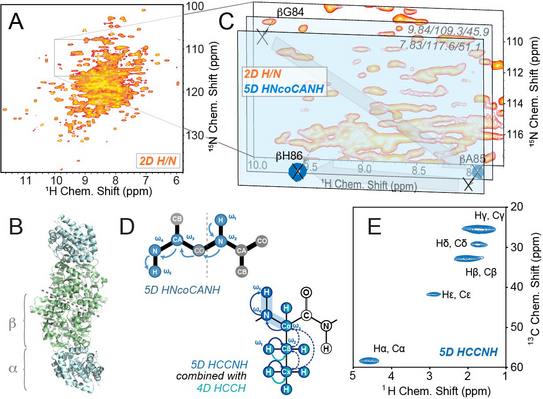
Die Gruppe um Prof. Rasmus Linser forscht an neuen Methoden und Anwendungen für die protonendetektierte Festkörper-NMR, um neue Einblicke in Proteine zu gewinnen, die bisher zu groß und komplex zu analysieren oder aus anderen Gründen nur schwer mit Lösungs-NMR zu untersuchen sind. Ein Hauptaugenmerk liegt dabei auf neuen, hochdimensionalen NMR Experimenten mit vier und fünf Dimensionen in Kombination mit „magic angle spinning“ (MAS), bei dem die Proteinprobe in einem kleinen Rotor mit Frequenzen von bis zu 100 Rotationen pro Sekunde im Magneten gedreht wird (Abb. 1). So können Signalüberlagerungen minimiert werden und letztendlich immer komplexere Systeme analysiert werden.
In einem aktuellen Forschungsartikel im renommierten Journal PNAS zeigen die Forscher, wie vier- und fünfdimensionale NMR Experimente dazu genutzt werden können, um neue Einblicke in das 2 x 72 kDa große, PLP-abhängige Enzym Tryptophan Synthase zu erlangen (Abb. 2A-D). Damit stellen sie einen neuen „Weltrekord“ für die Komplexität (d. h. Molekulargewicht der asymmetrischen Einheit) von Festkörper-NMR-Targets auf und überbieten deutlich die bisherige Bestmarke von 42 kDa. PLP-abhängige Enzyme sind von Interesse als Drug targets, Tryptophan Synthase im Speziellen zudem als Biokatalysator in der Biotechnologie. Ihr Katalysemechanismus ist jedoch auch nach Jahren intensiver Forschung noch nicht vollständig verstanden, auch dadurch, dass NMR Studien bisher durch das hohe Molekulargewicht stark limitiert gewesen sind.
In einer technischen Folgearbeit, just accepted in J. Phys. Chem. Lett., zeigt die Gruppe außerdem, wie fünf- und vierdimensionale Experimente verschachtelt werden können und so eine fast vollständige Signalzuordnung von allen Atomen in einem Modelprotein mit anschließender Strukturrechnung in nur wenigen Tagen erfolgen kann (Fig. 2E).
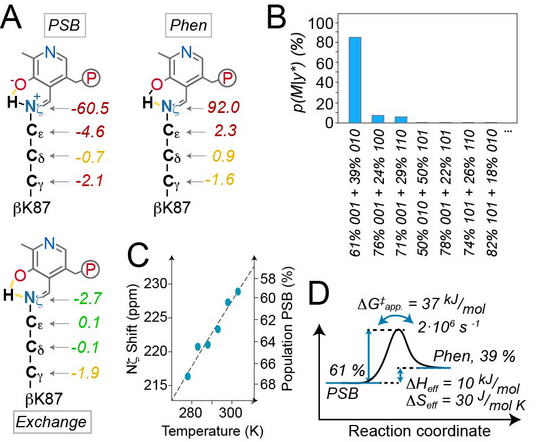
Die neu entwickelten, hochdimensionalen NMR Experimente konnten nun dazu beitragen, Signale zum ersten Mal eindeutig den einzelnen Aminosäuren zuzuordnen. In Kollaboration mit der Gruppe um Prof. Leonard Mueller von der UC Riverside (Density Functional Theory Rechnungen von erwarteten chemischen Verschiebungen) konnten die Autoren auf dieser Grundlage zeigen, dass verschiedene Konformationen des aktiven Zentrums der Β-Untereinheit das Tautomerisierungsgleichgewicht der Schiffbase (Abb. 3) modulieren, das bislang erste Indiz dafür, dass die allosterische Regulation des Enzyms auch die chemischen Eigenschaften der Reste in der aktiven Tasche, genauer den lokalen pKA der prosthetischen Gruppe, beeinflusst. Sie legen so außerdem den Grundstein für weitere NMR-basierte Studien an Tryptophan Synthase und anderen PLP-abhängigen Enzymen, welche die Dynamik dieser komplexen Enzyme genauer unter die Lupe nehmen.
Die Arbeiten an der TU Dortmund wurden im Rahmen des Emmy Noether-Programms und des Exzellenzclusters RESOLV durch die Deutsche Forschungsgemeinschaft (DFG) gefördert.
A. Klein, P. Rovó, V. V. Sakhrani, Y. Wang, J.B. Holmes, V. Liu, P. Skowronek, L. Kukuk, S. K. Vasa, P. Güntert, L. J. Mueller, R. Linser,
Atomic-resolution chemical characterization of (2x)72-kDa tryptophan synthase via four- and five-dimensional 1H-detected solid-state NMR,
Proc. Natl. Acad. Sci. U.S.A., 2022, 119 (4), e2114690119
A. Klein, S. K. Vasa, B. Söldner, K. Grohe, R. Linser,
Unambiguous Sidechain Assignments for Solid-State NMR Structure Elucidation of Non-deuterated Proteins via a Combined 5D/4D Sidechain-to-Backbone Experiment
J. Phys. Chem. Lett., 2022