Optische Kontrolle der Transkription von DNA
- Neues aus der Fakultät 2019
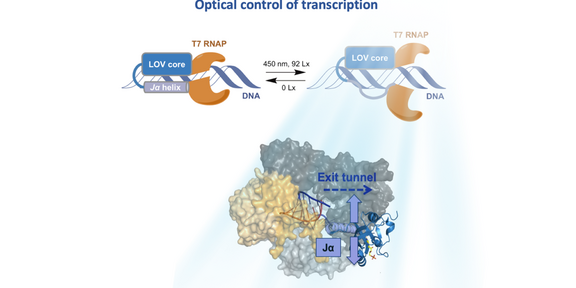
Natürliche, auf Licht reagierende Proteine ändern oftmals ihre drei-dimensionale Struktur, wenn sie mit Licht bestrahlt werden. Sind diese Proteine mit einem weiteren Protein, z. B. einem Enzym, kovalent verbunden, kann deren Aktivität durch die Änderung der Gesamtfaltung beeinflusst werden. Da Licht zudem mit hoher Präzision eingesetzt werden kann, sind Licht-schaltbare Proteine zunehmend von Interesse für die biochemische und medizinische Forschung.
Susanne Brakmann und Mitarbeiter/innen haben untersucht, wie die durch Licht ansprechbare „light-oxygen-voltage“-(LOV)-Proteindomäne so mit der RNA-Polymerase des Bakteriophagen T7 (T7 RNAP) kovalent verknüpft werden kann, dass die Aktivität des Enzyms durch Bestrahlung mit Licht gezielt steuerbar wird. Die Lichtschaltbarkeit der LOV-Domäne beruht auf der Entfaltung eines helicalen Abschnitts (Ja; im Bild lavendelfarben). Diese kleine Strukturänderung wird an die Polymerase (im Bild gelb/grau) weitergegeben und beeinflusst dort die Wechselwirkungen mit den Substraten (DNA, NTPs) und dem Produkt (RNA).
Das Team fand heraus, dass die LOV-Domäne unter (weitgehendem) Erhalt der Enzymaktivität an verschiedenen Positionen der T7 RNAP insertiert werden kann. Je nach Insertionsposition konnte die Aktivität durch Bestrahlung mit blauem Licht (470 nm) entweder erhöht oder erniedrigt werden. – Die neuen Polymerasevarianten sind nützliche Werkzeuge für die Untersuchung von RNA und DNA in zellulären und krankheitsbezogenen Prozessen.





