Das fehlende Fragment
- Neues aus der Fakultät 2024

Proteine erledigen den größten Teil der „To-dos“ innerhalb der Zelle, weshalb sie engmaschig und vielfältig durch Interaktionspartner reguliert werden. Um Regelsignale „von der Rückseite“ des Proteins kommend (allosterisch) einzubringen, nutzen Proteine dynamische Netzwerke, die Bindung von Interaktionspartnern in geänderte Beweglichkeitsverteilungen des Proteins übersetzen. In der in der „Angewandten“ erschienenen Arbeit zeigen Sara Medina Gomez (Experimentelles), Tye Gonzalez (Simulation) und Suresh Vasa (technische Expertise) aus dem AK Linser, dass solche Netzwerke durch intermolekularen Zusammenschluss erzeugt werden können.
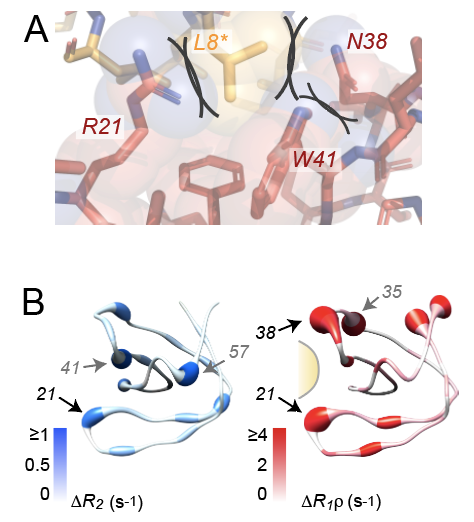
Die Bewegungen einer einzelnen Aminosäure, ihrer Seitenkette, ihres Rückgratanteils oder ganzer Domänen, sind durch sterische Restriktion und davon abgeleitete Aktivierungsbarrieren vorgegeben. Diese Rahmenbedingungen, die für die jeweilige atomare Dynamik maßgeblich sind, sind aber zeitlich stark variabel. So können die Zustände der umgebenden strukturellen Elemente (z. B. beengend oder Abstand haltend) die Beweglichkeit eines ersten Elementes mitbeeinflussen, und Ketten von voneinander abhängigen Beweglichkeiten, sogenannte dynamische Netzwerke, entstehen.
Während solche intramolekularen Netzwerke bekannt sind und vielfacht untersucht wurden (Noch immer fällt es dem Menschen nicht leicht, die Zusammenhänge gänzlich zu durchschauen…), sind intermolekulare Netzwerke wahrscheinlich aber noch nicht beschrieben worden. Das Wichtige, wenn es diese gäbe, wäre, dass sie temporär gebildet werden würden, wenn ein Interaktionspartner bindet, sie sich aber in Luft auflösten, sobald der Interaktionspartner abdissoziiert. Entsprechend würde eine Signalweiterleitung und entsprechende Regelmechanismen für Proteinfunktionalität durch die Bindung an- und durch ein Ablösen ausgeschaltet werden.
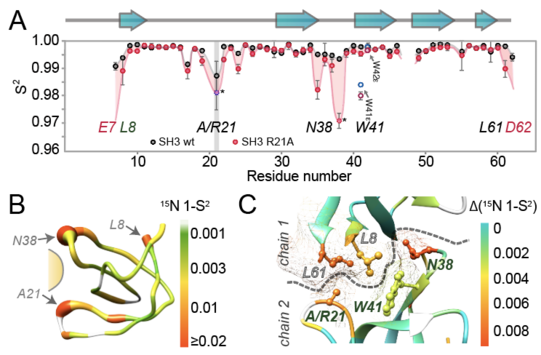
Dass die Formierung solcher Netzwerke und damit die Möglichkeit eines entsprechenden Regelprinzips prinzipiell möglich ist, zeigte die Gruppe anhand einer künstlichen Protein-Protein-Interaktion, die durch Kristallisation des Proteins kreiert wird, in Lösung (monomeres Protein) aber nicht vorhanden ist. Um die Kopplung zwischen zwei gegenüberliegenden Seiten der das zusätzliche Proteinsegment bindenden Tasche zu eruieren, wurden Punktmutation einzelner Aminosäuren eingeführt. Während die Mutationen in Lösung – ohne das verbindende Fragment – keinen Einfluss auf die gegenüberliegende Taschenseite hatte, fand im kristallinen Zustand (mit eingebettetem Bindeglied) eine starke Veränderung der Dynamik nicht nur an der Mutationsstelle statt, sondern auch auf der gegenüberliegenden Seite der Tasche.
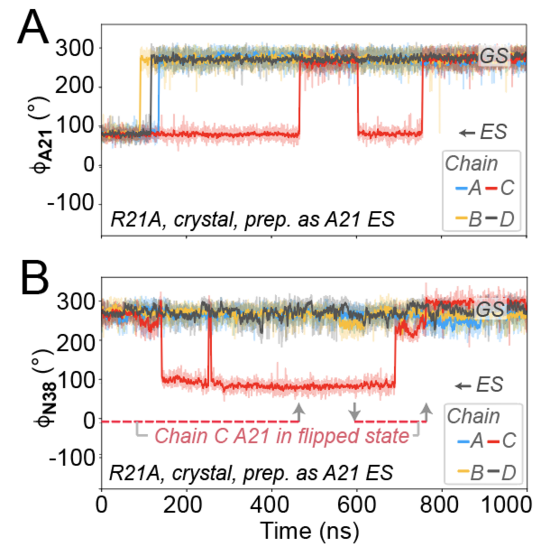
Da die experimentell beobachtete Kopplung auch einfach durch „regional“ veränderte Platzverhältnisse durch Mutation entstehen könnte, kreierte Tye Gonzalez MD Simulationen, in denen er die gegenseitigen Abhängigkeiten der Bewegungen zeitaufgelöst beobachten konnte. Und in der Tat konnte die Sequenz gekoppelter Ereignisse bestätigt werden: Ein Wegbiegen einer ersten Aminosäure erlaubt eine Beweglichkeit einer zweiten, wodurch mehr Platz für die dritte entsteht, die dadurch Bewegung zeigen kann, die ausbleibt, wenn die erste sich nicht gerade wegdreht. Da die beobachtete Verkettung dynamischer Aspekte vom mittleren, durch intermolekulare Assoziation erst eingebrachten Rest abhängt, scheint die Möglichkeit intermolekular gebildeter (und wieder auseinanderzunehmender) Regelnetzwerke tatsächlich zu existieren. Das Verständnis der intermolekularen Kopplung könnte helfen, die Mechanismen der sogenannten Allosterie (also der Regelung von Proteinfunktionalität von entfernten Stellen aus) besser zu verstehen und, z. B. pharmakologisch, zu nutzen.
S. Medina Gomez, T. Gonzalez, S. K. Vasa, R. Linser,
"Allostery at a protein-protein interface harboring an intermolecular dynamic network",
Angew. Chem., Int. Ed. (2024); DOI: 10.1002/anie.202411472






