Startpunkt für die Entwicklung von Wirkstoffen gegen einen tumorrelevanten zellulären Mechanismus entdeckt
- Neues aus der Fakultät 2020
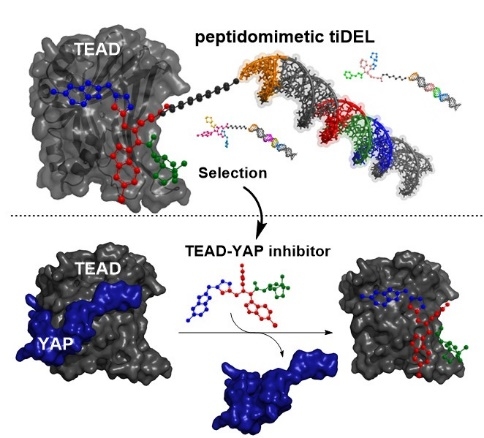
Eine neue Molekülklasse, welche in einen für viele Tumore essentiellen zellulären Mechanismus eingreift, konnte von Verena Kunig aus dem Arbeitskreis Brunschweiger in einer interdisziplinären und internationalen Forschungsarbeit gefunden werden. Die Moleküle hemmen die Bindung zweier Transkriptionsfaktoren, deren Interaktion von Tumorzellen genutzt wird, für das Zellwachstum, die Metastasenbildung und die Abwehr von Tumormedikamenten wichtige Prozesse in Gang zu bringen. Ihre Arbeit wurde kürzlich als VIP-Artikel in der renommierten Fachzeitschrift Angewandte Chemie einer internationalen Leserschaft vorgestellt. Lediglich 5 % aller Artikel erhalten den Status als VIP-Artikel, der als besonders wichtig eingeschätzten Arbeiten verliehen wird.
Die DNA-kodierte Substanzbibliothek wurde mittels der vom Arbeitskreis Brunschweiger in Dortmund entwickelten TiDEC-Technologie ausgehend von einem chemisch stabilen Hexathymidin („hexT“) -Adapteroligonukleotid hergestellt. Sie wurde von der Erstautorin unter Verwendung der Ugi-Mehrkomponentenreaktion um Indol-Strukturen zur Nachahmung der Tryptophan-Aminosäureseitenkette und damit zur Adressierung von Protein-Protein-Interaktionen entworfen. Durch anschließende Kupfer-vermittelte Alkin-Azid-Cycloaddition konnte eine Substanzbibliothek, bestehend aus Tausenden von DNA-kodierten Molekülen, generiert werden. Selektionsexperimente der DNA-kodierten Substanzbibliothek auf den krankheitsrelevanten Proteinen MDM2 und TEAD4 führten zur Identifizierung eines neuen MDM2-Binders sowie zur Entdeckung eines Inhibitors der Protein-Protein-Interaktion des Transkriptionsfaktors TEAD4 mit dem Co-Aktivatorprotein YAP. Diese Protein-Protein-Wechselwirkung ist fehlreguliert in vielen Tumoren, zum Beispiel durch Mutationen, die zu einer vermehrten Aktivierung des TEAD-YAP Transkriptionskomplexes führen. Die von Verena Kunig gefundene Molekülklasse hat das Potential, der Forschung auf diesem tumorrelevanten Mechanismus wichtige Impulse zu geben.
Die Publikation entstand in interdisziplinärer und internationaler Zusammenarbeit mit den Arbeitskreisen Rahnenführer und Fried aus der Fakultät Statistik der TU Dortmund, die einen neuen Algorithmus für die Wirkstoffsuche mit DNA-kodierten Molekülbibliotheken (DNA-encoded libraries, DELs) entwickelten; mit dem Arbeitskreis Dömling von der Universität Groningen, der beim Design der DEL wichtige Unterstützung gab; mit der Abteilung IV des Max-Planck-Instituts für molekulare Physiologie (MPI) und der AstraZeneca-MPI Satellite Unit, die die Wirkstoffe in vitro untersuchten; und mit der Lead Discovery Center GmbH Dortmund (LDC), sowie dem Arbeitskreis Ottmann von der Universität Eindhoven, welche die Moleküle in Tumorzellen untersuchten. Die Arbeit wurde in wichtigen Teilen vom Drug Discovery Hub Dortmund (DDHD) ermöglicht.





