Finetuning von Proteinplastizität via Directed Evolution
- Neues aus der Fakultät 2023

In Zusammenarbeit mit den Gruppen um Prof. Daniel Summerer und Prof. Lars Schäfer von der RUB zeigen Dr. H. Singh und Prof. R. Linser im Journal NAR, wie die Directed Evolution eines Proteins eine maßgeschneiderte Veränderung der Proteinplastizität hervorbringt, die das selektive Erkennen des intendierten Bindungspartners ermöglicht. Es überrascht niemanden, dass es die gerichtete Evolution fertigbringt, oberflächliche Aminosäuren so zu verändern, dass sie zielgenau auf neue Interaktionspartner zugeschnitten sind. Wie verhält es sich aber mit der wichtigen Eigenschaft der Moleküldynamik? Ist die ein Zufallskonstrukt oder gibt es Evolution nicht nur von Struktur, sondern auch von Plastizität?
Für ihre Funktionalität benötigen Proteine nicht nur eine (meist) wohl-definierte dreidimensionale Struktur, sondern auch eine genau dazu passende Beweglichkeit. Trotz dieses Bewusstseins wundert es Biologen häufig, dass die gerichtete Evolution von Enzymen (z. B. für die Biotechnologie) nicht nur Aminosäuren zielführend verändert, die in direktem Kontakt zu Substraten und deren Übergangszuständen stehen, sondern auch solche, die weit entfernt von jeglichen Interaktionen liegen. Der Grund liegt auf der Hand: Neben Interaktionsflächen muss auch die Plastizität des Proteins (ortsaufgelöst) auf genau das zugeschnitten werden, was von dem Enzym verlangt wird. Und die Beweglichkeit verändernde Aminosäuren liegen nun einmal nicht notwendigerweise dort, wo man sie vermutet.
Genau dies konnten Dr. Himanshu Singh und Kollegen nur für einen sogenannten Epigenetischen Leser (Epigenetic Reader) zeigen. Dieses Protein wurde von der Gruppe Summerer mittels gerichteter Evolution auf die zielgerichtet Erkennung bestimmter epigenetischer Basen „evolviert“, um irgendwann einmal als molekulare Tool zur Erkennung und zum Verständnis der Epigenetik zu fungieren, also der kovalenten Modifikationen von Nukleinsäuren und anderer Maschinerie, die unterschiedliche Phänotypen aus derselben genetischen Information zu generieren vermag. Auch hier wurden von der Directed Evolution Veränderungen von Aminosäureresten als notwendig aufgezeigt, die weit von der Protein-DNA Interaktionsfläche entfernt liegen. Diese Veränderungen tief im hydrophoben Kern des Proteins schienen zuerst zufällig und unerklärlich. Detaillierte Studien zur Proteindynamik mittels biomolekularer NMR-Spektroskopie sowie Molekulardynamik-(MD-)simulationen zeigten dann allerdings, dass Mutationen in diesem Bereich das Zusammenhängen der einzelnen Sekundärstrukturelemente stärken oder schwächen und damit die Gesamtplastizität des Proteins und seine Bindungskompetenz feintunen. Zwar haben Proteinvarianten ohne die Kernmutation zwar das richte Bindungsinterface, können aber trotzdem nicht mit hoher Affinität binden. Erst das richtige Maß an Plastizität ermöglicht es dem Readerprotein, sein Target selektiv zu erfassen, und so sind auch diese Mutationen für die Funktionalität des molekularen Tools notwendig.
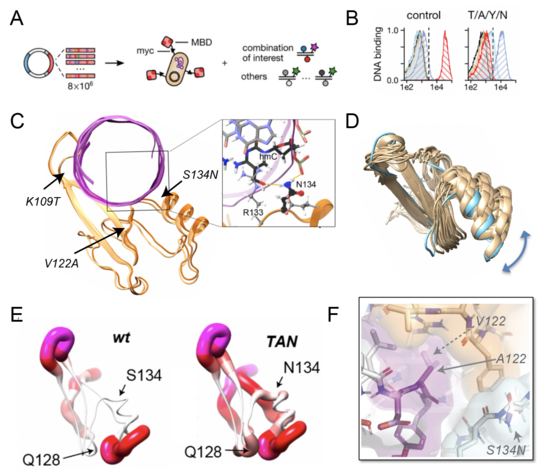
Die Bedeutung der Beweglichkeit der Moleküle, die weit schwerer zu fassen ist als ihre dreidimensionale Struktur, gerät leicht aus dem Augenmerk der Strukturbiologie und Strukturchemie. Die vorliegende Studie beweist allerdings, dass sowohl die Natur als auch die künstliche Evolution von Proteinen im Labor in der Lage sind, notwendige Beweglichkeitseigenschaften genau so zu variieren, bis die gewünschte Funktionalität erhalten wird.
Der Artikel in der Zeitschrift Nucleic Acids Research (NAR) wurde im Juli als Coverstory veröffentlicht.
H. Singh, C. K. Das, B. Buchmuller, L. Schäfer*, D. Summerer*, R. Linser*,
Epigenetic CpG Duplex Marks Probed by an Evolved DNA Reader via a Well-Tempered Conformational Plasticity
Nucleic Acids Res., 2023, 51, 6495–6506, DOI: 10.1093/nar/gkad134





